Technologie : Les fameux CNN sont poussés vers la sortie par de nouvelles approches qui permettent de mieux comprendre les objets de recherche autour de l’Intelligence Artificielle. Analyse des derniers travaux de Google en la matière.
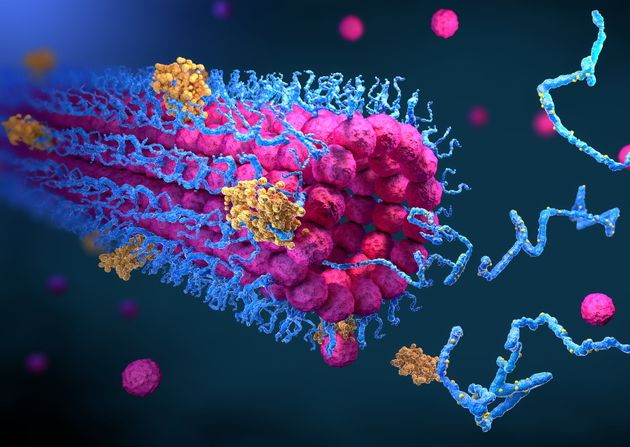
DeepMind, l’unité d’IA de Google qui a inventé le réseau neuronal Alpha Zero, champion d’échecs il y a quelques années, a de nouveau surpris en novembre dernier avec un programme qui a résolu un problème vieux de plusieurs décennies. A savoir comment les protéines se replient. Le programme a largement battu tous ses concurrents, ce qu’un chercheur a appelé un « moment décisif », qui promet de révolutionner la biologie.
AlphaFold 2 n’a été décrit à l’époque qu’en termes brefs, dans un billet de blog de DeepMind et dans un résumé d’articles fournis par DeepMind pour la compétition à laquelle ils ont soumis le programme. Il s’agissait de la compétition bisannuelle Critical Assessment of Techniques for Protein Structure Prediction.
En juillet dernier, DeepMind a finalement révélé la façon dont il a procédé, en publiant non seulement un billet de blog, mais aussi un article de 16 pages, rédigé par John Jumper et ses collègues de DeepMind, dans le magazine Nature, une collection de 62 pages de matériel supplémentaire et une bibliothèque de codes sur GitHub. Dans un article présentant de nouveaux détails, Ewan Calloway de Nature qualifie le transfert de données de « structure protéique accessible au plus grand nombre ».
Les convolutions ne sont plus de mise et les graphes sont de retour
Alors, qu’avons-nous appris ? Quelques éléments. Comme son nom l’indique, ce réseau neuronal est le successeur du premier AlphaFold, qui avait également battu ses concurrents lors du concours précédent, en 2018. La nouveauté la plus importante d’AlphaFold 2 est que faire des progrès en intelligence artificielle peut nécessiter ce que l’on appelle un changement d’architecture.
L’architecture d’un logiciel est l’ensemble spécifique d’opérations et la façon dont elles sont combinées. Le premier AlphaFold était constitué d’un réseau neuronal convolutif, ou « CNN » (convolutional neural network), un réseau neuronal classique qui a été le cheval de bataille de nombreuses percées en matière d’IA au cours de la dernière décennie.
Mais les convolutions ne sont plus de mise et les graphes sont de retour. Ou, plus précisément, la combinaison des réseaux de graphes avec ce que l’on appelle l’attention.
La partie d’Alpha Fold qui utilisait les convolutions est abandonnée dans Alpha Fold 2, remplacée par toute une série de mécanismes d’attention.
On parle de réseau de graphes lorsqu’une collection d’éléments peut être évaluée en fonction de leur parenté, comme les personnes abonnées à un réseau social et la façon dont elles sont liées par des liens d’amitié. Dans ce cas, AlphaFold utilise des informations sur les protéines pour construire un graphique de la proximité des différents acides aminés.
Ces graphes sont manipulés par le mécanisme d’attention qui a gagné en popularité dans de nombreux secteurs de l’IA. L’attention en matière d’Intelligence Artificielle consiste à ajouter une puissance de calcul supplémentaire à certains éléments de données d’entrée. Les programmes qui exploitent l’attention ont permis de réaliser des percées dans divers domaines, mais surtout dans le traitement du langage naturel, comme dans le cas du Transformer de Google.
Au final, la partie d’Alpha Fold qui utilisait les convolutions est abandonnée dans Alpha Fold 2, remplacée par toute une série de mécanismes d’attention.
L’attention conduit à la communication entre les parties du programme
L’utilisation de l’attention est présente tout au long d’AlphaFold 2. La première partie d’AlphaFold est ce que l’on appelle EvoFormer. Et elle utilise l’attention pour concentrer le traitement sur le calcul du graphique de la relation entre chaque acide aminé et un autre acide aminé. En raison des formes géométriques créées dans le graphique, Jumper et ses collègues qualifient cette opération d’estimation du graphique d' »auto-attention triangulaire ».
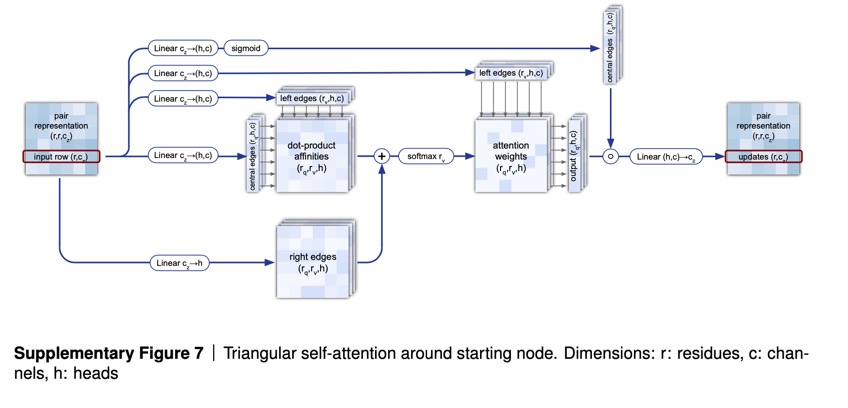
Faisant écho aux programmes de langage naturel, l’EvoFormer permet au triangle d’attention d’envoyer des informations vers les groupes de séquences d’acides aminés, connus sous le nom d' »alignements multi-séquences » ou « MSA » (multi-sequence alignments), un terme courant en bioinformatique dans lequel des séquences d’acides aminés apparentées sont comparées pièce par pièce.
Les auteurs considèrent que les MSA et les graphes sont en quelque sorte en conversation grâce à l’attention, ce qu’ils appellent un « encastrement conjoint » (joint embedding). Ainsi, l’attention conduit à la communication entre les parties du programme.
Pourquoi est-il important que les graphes, et l’attention, aient remplacé les convolutions ?
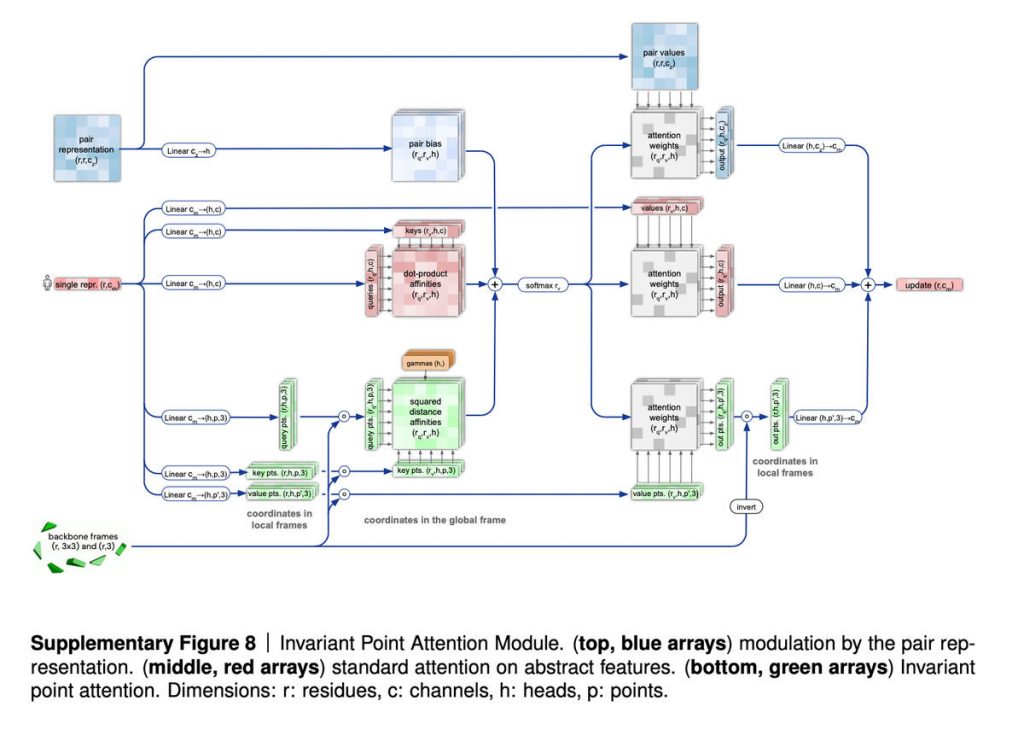
La deuxième partie d’AlphaFold 2, après l’EvoFormer, est ce qu’on appelle un module de structure (Structure Module), qui est censé prendre les graphes que l’EvoFormer a construit et les transformer en spécifications de la structure tridimensionnelle de la protéine, le résultat qui a remporté le concours CASP (Critical Assessment of Techniques for Protein Structure Prediction).
Ici, les auteurs ont introduit un mécanisme d’attention qui calcule les parties d’une protéine de manière isolée, appelé mécanisme d' »attention par points invariants ». Ils le décrivent comme « une opération d’attention tenant compte de la géométrie ».
Le module de structure initie les particules à une sorte de point d’origine dans l’espace, que vous pouvez considérer comme un champ de référence 3D, appelé « gaz de résidus », et procède ensuite à la rotation et au déplacement des particules pour produire la configuration 3D finale. Là encore, l’important est que les particules soient transformées indépendamment les unes des autres, en utilisant le mécanisme d’attention.
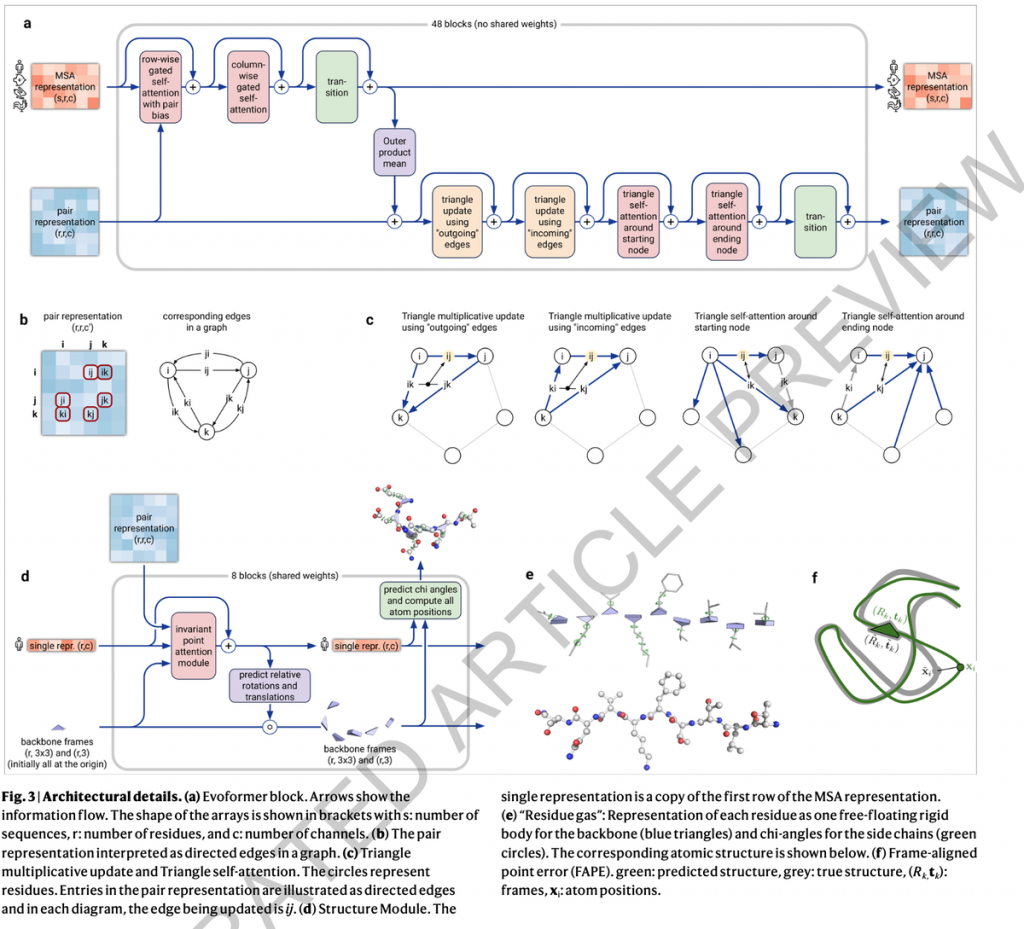
Pourquoi est-il important que les graphes, et l’attention, aient remplacé les convolutions ? Dans le résumé proposé pour la recherche l’année dernière, Jumper et ses collègues ont souligné la nécessité de dépasser la fixation sur les structures dites « locales ».
Un réseau neuronal a vraiment besoin de revoir constamment ses prédictions
Pour en revenir à AlphaFold 1, le réseau neuronal convolutif fonctionnait en mesurant la distance entre les acides aminés, puis en résumant ces mesures pour toutes les paires d’acides aminés sous la forme d’une image en deux dimensions, appelée histogramme de distance ou « distogramme ». Le réseau neuronal convolutionnel fonctionne ensuite en examinant cette image pour trouver des motifs locaux qui se transforment en motifs de plus en plus larges couvrant toute la gamme des distances.
Mais cette progression ordonnée à partir de motifs locaux peut ignorer les dépendances à longue portée, qui sont l’un des éléments importants que l’attention est censée capter. Par exemple, le mécanisme d’attention de l’EvoFormer peut relier ce qui est appris dans le mécanisme du triangle d’attention à ce qui est appris dans la recherche de l’ASM (multi-sequence alignments). Mais pas seulement une section de la MSA, mais l’univers entier des séquences d’acides aminés apparentées.
L’attention permet donc de faire des bonds en avant qui sont plus « globaux ».
Une autre chose que nous voyons dans AlphaFold est l’objectif de bout en bout. Dans la version originale d’AlphaFold, l’assemblage final de la structure physique était simplement guidé par les circonvolutions et ce qu’elles produisaient. Dans AlphaFold 2, Jumper et ses collègues ont mis l’accent sur la formation du réseau neuronal de « bout en bout ».
Par conséquent, un autre élément important à retenir d’AlphaFold 2 est la notion qu’un réseau neuronal a vraiment besoin de revoir constamment ses prédictions. Cela est vrai à la fois pour l’opération de recyclage, mais aussi à d’autres égards. Par exemple, l’EvoFormer, qui crée les graphiques des acides aminés, révise ces graphiques à chacune des multiples étapes, appelées « blocs », de l’EvoFormer. Jumper et son équipe qualifient ces mises à jour constantes de « communication constante » à travers le réseau.
Le mystère du pliage des protéines demeure
Comme le notent les auteurs, grâce à cette révision constante, la partie Structure du programme semble affiner « en douceur » ses modèles de protéines. « AlphaFold apporte des améliorations incrémentielles constantes à la structure jusqu’à ce qu’il ne puisse plus s’améliorer », écrivent-ils. Parfois, ce processus est « gourmand », c’est-à-dire que le module Structure trouve une bonne solution au début de ses couches de traitement, parfois il prend plus de temps.
Quoi qu’il en soit, les avantages de la formation d’un réseau neuronal, ou d’une combinaison de réseaux, dans ce cas, semblent devoir être un point d’intérêt pour de nombreux chercheurs.
Parallèlement à cette grande leçon, un mystère important reste au centre d’AlphaFold 2 : pourquoi ?
Pourquoi les protéines se replient-elles de la manière dont elles le font ? AlphaFold 2 a ouvert la perspective de révéler la structure de toutes les protéines de l’univers. Mais AlphaFold 2 n’explique pas pourquoi les protéines prennent la forme qu’elles ont.
Les protéines sont des acides aminés, et les forces qui les font s’enrouler dans une forme donnée sont assez simples, des choses comme certains acides aminés étant attirés ou repoussés par des charges positives ou négatives, et certains acides aminés étant « hydrophobes », ce qui signifie qu’ils restent plus éloignés des molécules d’eau.
Ce qui manque encore, c’est une explication de la raison pour laquelle certains acides aminés prennent des formes si difficiles à prévoir.
AlphaFold 2 est une réalisation étonnante en termes de construction d’une machine permettant de transformer des données de séquence en modèles de protéines, mais nous devrons peut-être attendre une étude plus approfondie du programme lui-même pour savoir ce qu’il nous dit sur le tableau d’ensemble du comportement des protéines.
Source : ZDNet.com